肿瘤基因如何检测?常见基因检测技术有FISH、PCR、Sanger测序和NGS。NGS广泛用于复杂疾病、遗传病筛查、肿瘤评估等。肿瘤Panel检测基于高通量测序,周期短、通量高、覆盖全面、成本低,助力精准诊断。定期监测变异,调整方案,为临床试验和跨适应症用药提供依据。NGS大panel结合其他靶标,是肿瘤免疫治疗标志物检测利器。但存在基因覆盖不全和测序深度不足等挑战,可能影响变异检测的准确性。大panel检测虽全面但成本高,需权衡利弊。
自适应测序技术,作为纳米孔测序平台所独有的特色,依托软件控制的精密机制,实现了对测序序列的高效富集。其核心运作原理在于,依据用户提供的特定序列信息,智能判断并决定是否对目标序列进行详尽的测序操作或适时弹出(如图1所示)。在操作过程中,系统会细致检查DNA分子穿越纳米孔时最初几百个碱基的序列,并将其与预先设定的靶向区域进行精确比对。基于这一比对结果,软件将迅速作出判断:若该核酸分子恰好符合用户的目标序列要求,则立即启动全面测序流程;反之,若不匹配,则通过巧妙调整纳米孔两端的电压,将该分子迅速而优雅地弹出,从而为后续新的核酸分子腾出宝贵的纳米孔空间。尤为值得一提的是,自适应测序技术极大地简化了研究流程,研究者无需再耗费大量时间与精力进行繁琐的前期样品准备或富集工作,即可直接投身于激动人心的测序过程之中。
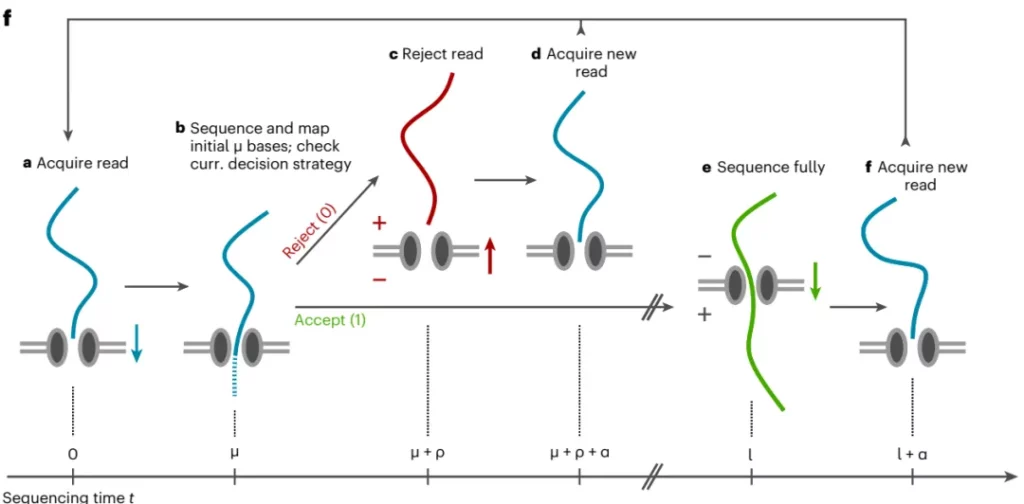
图1:纳米孔自适应测序流程
✔ 无需PCR扩增,减少了扩增偏差,同时保留了碱基和修饰信息。
✔ 提供富集和耗尽两种模式,生信分析轻松富集目标区域,无需复杂实验。
✔ 快速建库,最快10分钟完成,适合临床快速应用。
✔ 长读长特性跨越大片段区域,提升复杂结构变异检测能力。
✔ 实时数据分析直接富集DNA目标序列,无需额外步骤。
✔ 经济高效筛选已知和新的变异,适用于大型目标区域靶向测序。
1. 文库构建原理
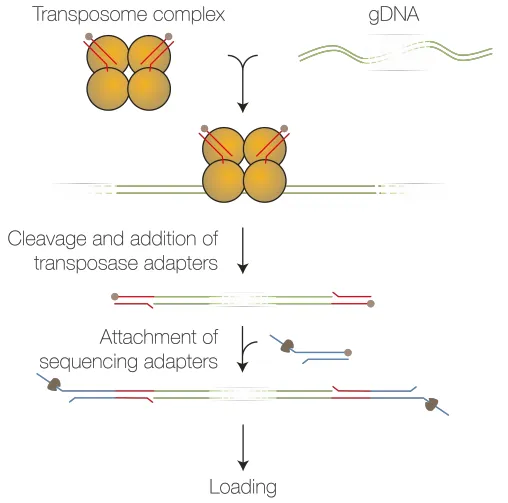
图2:ONT-DNA-RRMS文库构建和测序流程图
首先提取基因组DNA样本,对DNA进行Tn5片段化,打断后的DNA连接测序接头。在ONT测序仪器上面选择设计好的针对特定检测基因的bed文件,进行自适应采样测序程序。
2. 技术路线

案例1
中文标题:利用靶向自适应采样长读长测序对儿童白血病进行基因组分析
期刊:Blood Cancer Journal
影响因子:12.9
样本:28名白血病儿童(10名急性髓性白血病,13名B细胞急性淋巴细胞白血病,5名T细胞ALL)
技术方法:Targeted adaptive sampling long-read sequencing
靶向测序区域:包括466个与血液恶性肿瘤相关的基因
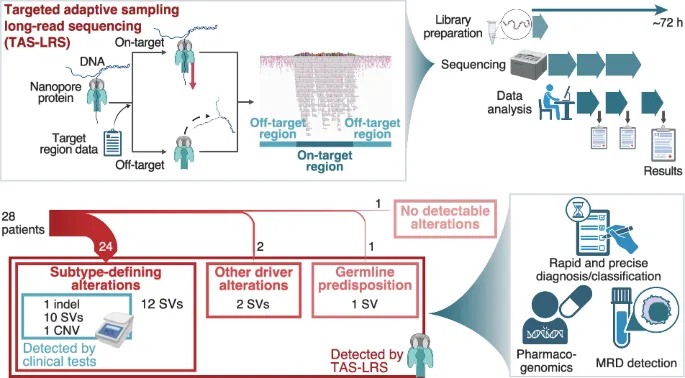
纳米孔测序仪是长读数测序仪,具自适应采样功能,无需额外制备,覆盖广泛靶区域,在SV检测和单倍型变异识别上表现出色,且文库制备和测序时间短于传统方法。鉴定出498个SNV、35个小插入缺失和632个SVs,其中部分被认为是驱动改变。在24名患者中,TAS-LRS确定了85.7%的基因组亚型,并发现新变异体均为SVs。长读长结合靶向捕获测序能迅速鉴定多峰基因变体,包括融合基因SVs、隐性易位和复杂SVs,低覆盖率读取数据可确定CNV,覆盖大至染色体小至几个基因。总之,TAS-LRS通过纳米孔测序快速有效分析儿科白血病基因组,利于鉴定SVs和CNVs。其应用将改进白血病治疗实践。
案例2
中文标题:通过遗传性癌症患者基因组评估目标自适应采样长读长测序
期刊:npj Genomic Medicine
影响因子:4.7
样本:33例疑似遗传癌综合征患者
技术方法:Targeted adaptive sampling long-read sequencing
靶向测序区域:147个癌症易感基因
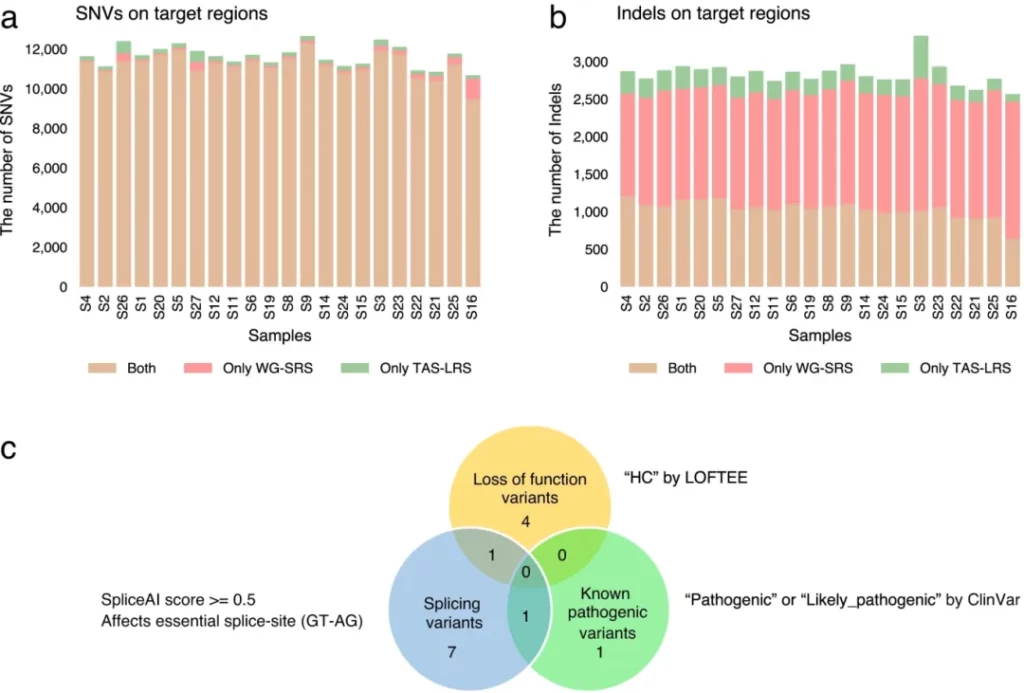
作者开发了TAS-LRS计算工作流程,应用于疑似遗传性癌症患者的基因组评估,识别单核苷酸变异并阐明结构变异形式。新鉴定了家族性腺瘤性息肉病患者的SVA元件及其起源位点。我们还证明,自适应采样的脱靶读数可用于SNP基因分型,计算多基因风险评分。在林奇综合征患者中发现等位基因特异性MLH1启动子高甲基化。TAS-LRS工作流程能同时捕获单基因、多基因风险变异及表观遗传改变,成为遗传病研究和诊断的有效平台。
案例3
中文标题:纳米孔自适应采样检测核苷酸变异和改善大规模重排特征诊断癌症易感性
期刊:Cancer Research
影响因子:12.5
样本:30份临床种系样本携带已知BRCA1、BRCA2、PALB2和MLH1基因改变
技术方法:Targeted adaptive sampling long-read sequencing
靶向测序区域:152个癌症易感基因
队列中11个样本有大规模重组(LSR),由ONT测序检测。除检测到LSR位点外,发现BRCA1基因中外显子13的MLPA扩增实为外显子12和13的扩增,在患者亲属中亦证实。另一LSR为BRCA1基因完全缺失,由近140 kbp大量缺失导致,包含5种基因。ONT测序在低覆盖率样本中检测到5类SNV。生物信息学检测确认均聚物区域3个突变。分析完整基因时,发现新未知变异。通过Sanger测序验证8个新变异,均证实,表明ONT检测变异可靠。总之,ONT自适应采样测序适用于种系改变分析,改善LSR特性,低覆盖率下可检测SNV。
1. Ahsan, M.U., Gouru, A., Chan, J. et al. A signal processing and deep learning framework for methylation detection using Oxford Nanopore sequencing. Nat Commun 15, 1448 (2024). https://doi.org/10.1038/s41467-024-45778-y
2. Kato, S., Sato-Otsubo, A., Nakamura, W. et al. Genome profiling with targeted adaptive sampling long-read sequencing for pediatric leukemia. Blood Cancer J. 14, 145 (2024). https://doi.org/10.1038/s41408-024-01108-5
3. Nakamura, W., Hirata, M., Oda, S. et al. Assessing the efficacy of target adaptive sampling long-read sequencing through hereditary cancer patient genomes. npj Genom. Med. 9, 11 (2024). https://doi.org/10.1038/s41525-024-00394-z
4. Chevrier, Sandy, et al. “Nanopore adaptive sampling detects nucleotide variants and improves large scale rearrangement characterization for diagnosis of cancer predisposition.” Cancer Research 84.6_Supplement (2024): 2936-2936.
https://doi.org/10.1158/1538-7445.AM2024-2936

电话:027-870502099
邮箱:sales@rxbio.cc
地址:武汉市东湖高新区高新二路388 号
光谷生物医药加速器 18 栋 1-2层
单细胞多组学 空间转录组
三代测序 功能基因组
表观遗传学 互作组学
单细胞大数据 数据深度挖掘

欢迎关注公众号「瑞兴生物」