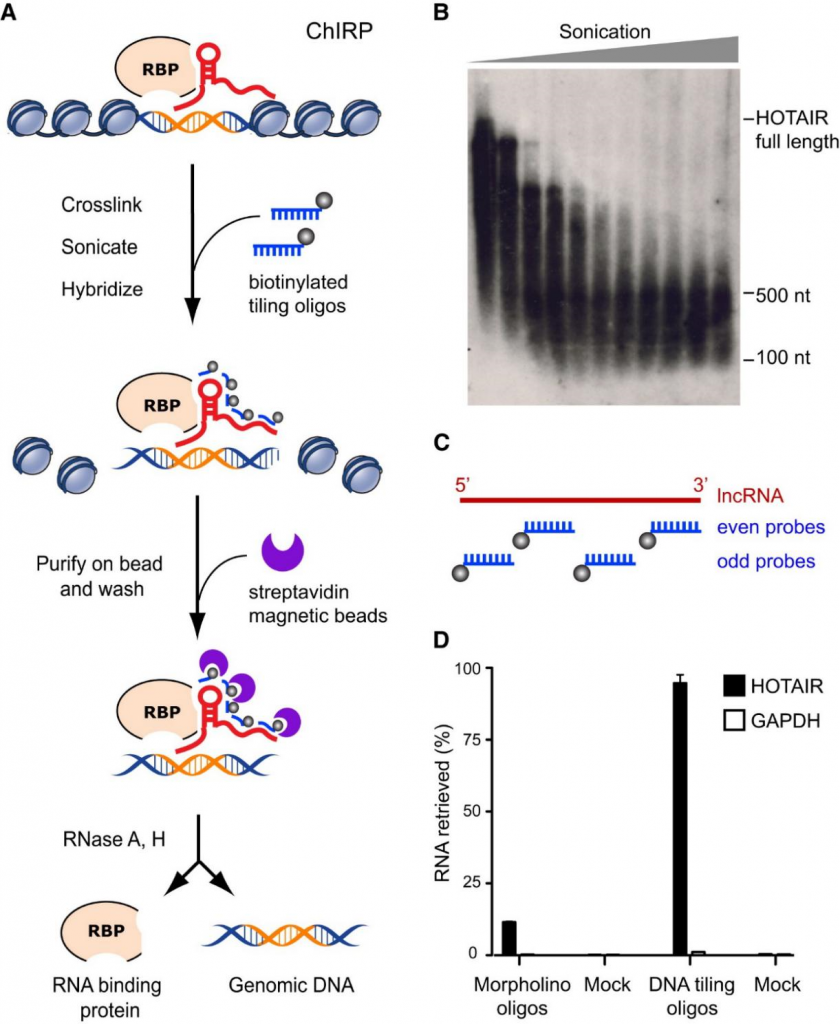
1.lncRNA的序列保守性比较差
2.空间折叠结构不太好预测
3.RNA和DNA的相互作用并不完全靠序列互补配对来完成的。
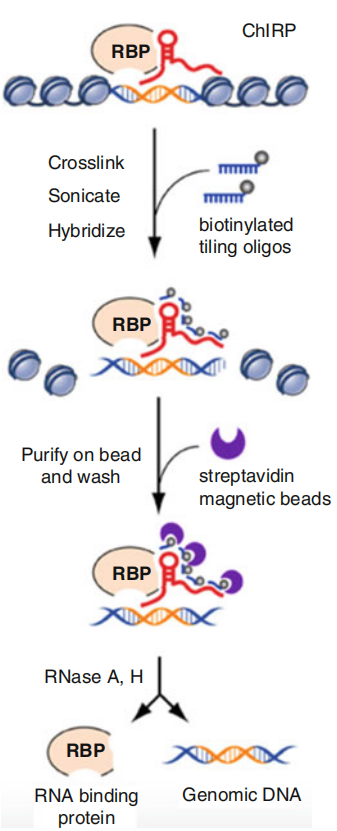
因此,lncRNA在核内转录调控机制的方法学从生物信息预测的角度来讲比较难以预测,ChIRP就是为解决此类需求而开发的实验方法。
为了有力地捕获lncRNA,ChIRP使用的探针是120-nt的长探针,长探针拥有降低背景噪音的优势。ChIRP技术不需要对染色质相互作用中涉及的lncRNA结合域有事先了解认知,在整个目标RNA上平铺寡聚核苷酸,使所有潜在的杂交位点都被充分利用,以确保即使在广泛的蛋白质- RNA相互作用、RNA二级结构或部分RNA降解的情况下也能对目标RNA及其结合的蛋白质和DNA进行捕获。
2. 收集细胞:收获用于ChIRP实验的细胞;
3. 对细胞进行交联,并收集细胞颗粒:用戊二醛交联收集的细胞,以保存RNA染色质相互作用,并制备细胞颗粒;
4. 细胞裂解:裂解交联细胞制备细胞裂解液;
5. 超声处理:通过超声处理交联细胞裂解物来剪切DNA;
6. ChIRP:将生物素化DNA探针与RNA杂交并分离结合染色质;
7. RNA分离:从ChIRP样本中提取RNA部分,通过RT-qPCR进行定量,以确定富集效率,并通过测序或定量PCR进行鉴定;
8. DNA分离:从ChIRP样本中提取DNA片段,通过测序或定量PCR进行鉴定;
9. 蛋白质分离:从ChIRP样本中提取蛋白质,通过质谱或WB实验进行鉴定。
细胞:≥300ul干体积
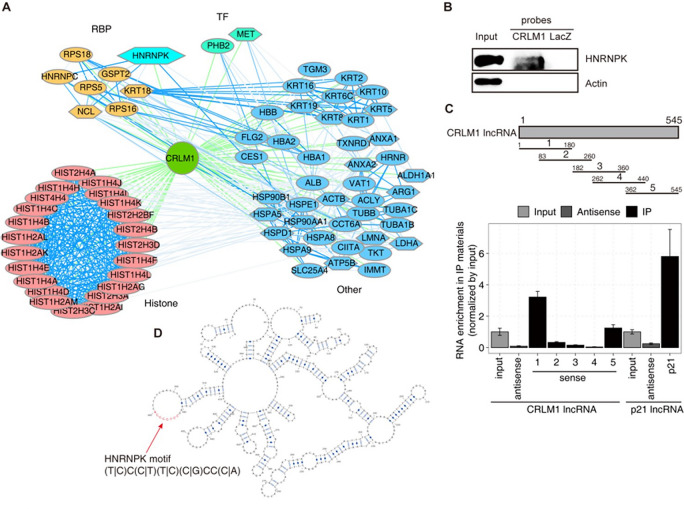
应用案例二——LncRNA CRLM1通过和HnRNPK互作来促进结直肠癌转移
Engreitz, J. M., K. Sirokman, P. McDonel, A. A. Shishkin, C. Surka, P. Russell, S. R. Grossman, A. Y. Chow, M. Guttman and E. S. Lander (2014). ``RNA-RNA interactions enable specific targeting of noncoding RNAs to nascent Pre-mRNAs and chromatin sites.`` Cell 159(1): 188-199.
Hacisuleyman, E., L. A. Goff, C. Trapnell, A. Williams, J. Henao-Mejia, L. Sun, P. McClanahan, D. G. Hendrickson, M. Sauvageau, D. R. Kelley, M. Morse, J. Engreitz, E. S. Lander, M. Guttman, H. F. Lodish, R. Flavell, A. Raj and J. L. Rinn (2014). ``Topological organization of multichromosomal regions by the long intergenic noncoding RNA Firre.`` Nat Struct Mol Biol 21(2): 198-206.
McHugh, C. A., C. K. Chen, A. Chow, C. F. Surka, C. Tran, P. McDonel, A. Pandya-Jones, M. Blanco, C. Burghard, A. Moradian, M. J. Sweredoski, A. A. Shishkin, J. Su, E. S. Lander, S. Hess, K. Plath and M. Guttman (2015). ``The Xist lncRNA interacts directly with SHARP to silence transcription through HDAC3.`` Nature 521(7551): 232-236.
Munschauer, M., C. T. Nguyen, K. Sirokman, C. R. Hartigan, L. Hogstrom, J. M. Engreitz, J. C. Ulirsch, C. P. Fulco, V. Subramanian, J. Chen, M. Schenone, M. Guttman, S. A. Carr and E. S. Lander (2018). ``The NORAD lncRNA assembles a topoisomerase complex critical for genome stability.`` Nature 561(7721): 132-136.
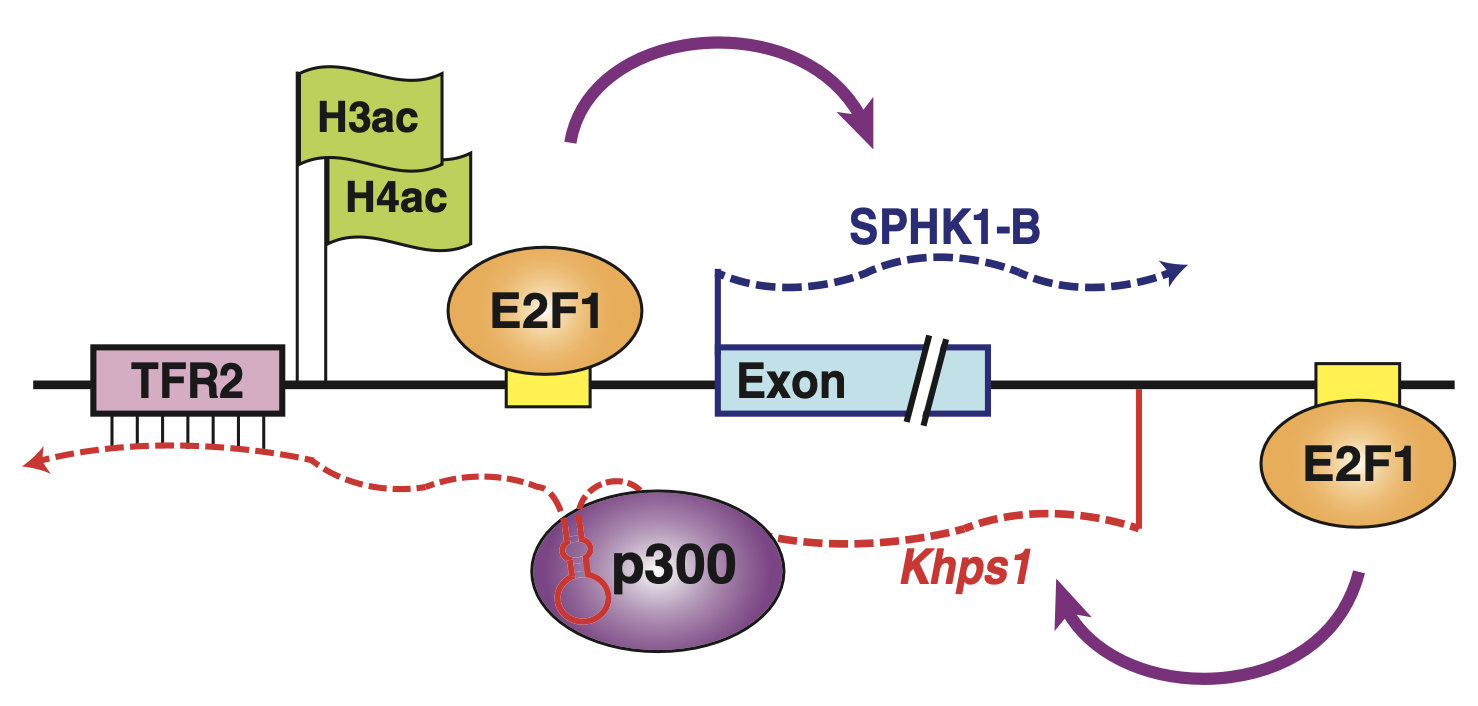
Postepska-Igielska, A., A. Giwojna, L. Gasri-Plotnitsky, N. Schmitt, A. Dold, D. Ginsberg and I. Grummt (2015). ``LncRNA Khps1 Regulates Expression of the Proto-oncogene SPHK1 via Triplex-Mediated Changes in Chromatin Structure.`` Mol Cell 60(4): 626-636.
Wang, Z., J. Chen, F. Sun, X. Zhao, Y. Dong, S. Yu, J. Li and H. Liang (2022). ``LncRNA CRLM1 inhibits apoptosis and promotes metastasis through transcriptional regulation cooperated with hnRNPK in colorectal cancer.`` Cell Biosci 12(1): 120.