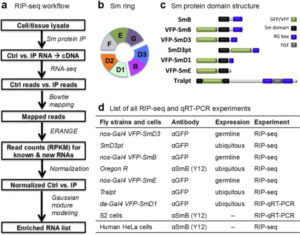
RIP-seq技术介绍
RIP技术(RNA Binding Protein Immunoprecipitation,RNA结合蛋白免疫沉淀),是研究细胞内RNA与蛋白结合情况的技术,是了解转录后调控网络动态过程的有力工 具,能获得RBP的调控靶点,同时也能帮助发现miRNA的调节靶点。RIP技术运用针对目标蛋白的抗体把相应的RNA-蛋白复合物沉淀下来,然后经过分离纯化就可以对结合在复合物上的RNA进行分析。RIP是染色质免疫沉淀ChIP技术的类似应用,但由于研究对象是RNA-蛋白复合物而不是DNA-蛋白复合物,RIP实验的优化条件与ChIP实验不太相同(如复合物不需要固定,RIP反应体系中的试剂和抗体绝对不能含有RNA酶,抗体需经RIP实验验证等)。RNA免疫共沉淀测序(RIP-seq),将RIP与新一代测序技术结合起来,在基因组范围内发现与蛋白或蛋白复合物相结合的RNA。
产品优势
RIP-seq研究的是自然状态下最原始的RBP和RNA的互作状态,捕获到的RNA未经酶切片段化,获得的RNA片段较长,也无需RBP切胶纯化步骤,操作流程简单,成功率高,但是不能得到RBP直接结合的RNA位点。
我司也自主研发了iRIP-seq(improved RIP-seq)技术,其整合了RIP-seq操作的简洁性和CLIP-seq识别RNA结合位点的优势,可以高效探测RBP直接和间接 的RNA 结合位点。在该项技术中,我们取消了凝胶纯化和 RNP 标记步骤,并保留了 CLIP操作的其他步骤,改进了文库构建的方法。
实验原理
·用抗体或表位标记物捕获细胞核内或细胞质中内源性的RNA结合蛋白。
·防止非特异性的RNA的结合。
·免疫沉淀把RNA结合蛋白及其结合的RNA一起分离出来。
·结合的RNA序列通过microarray(RIP-Chip),定量RT-PCR或高通量测序(RIP-Seq)方法来鉴定。
iRIP-seq实验操作流程
案例解析
案例1
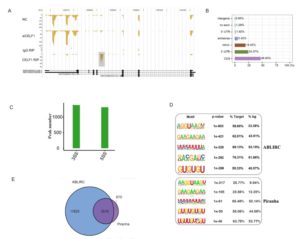
FOXN3是一种转录抑制因子,SIN3A是ER+细胞(雌激素受体阳性细胞)中的一种抑制因子复合物,本文作者通过研究FOXN3的病理学功能机制,发现FOXN3这种转录因子与SIN3A会发生蛋白质之间的互相作用,并且这种互作需要由乳腺癌细胞中的雌激素诱导产生的NEAT1(一种Long noncoding RNAs)的参与。为了研究介导FOXN3和SIN3A复合体互相作用的RNA,使用了iRIP-seq技术(生命之美自主开发的技术,基于RIP-seq的改进版)最终通过两种数据分析方法(Piranha和CIMS)筛选出两种lncRNA,NEAT1和MALAT1(图2A和2B),然后再通过对NEAT1和MALAT1敲除和过表达后进行CoIP检测的方法确定了NEAT1是FOXN3和SIN3A互作所必需的(图2C)。在小鼠体内实验证实FOXN3-NEAT1-SIN3A复合物促进癌细胞肺转移(图2D)。
FOXN3结合RNA的RIP-seq数据分析结果展示
FOXN3结合RNA的RIP-seq数据分析结果展示,及实验验证结果展示
案例解析
案例2
采用miR-31基因敲除(31-KO)和C57BL/6J野生型小鼠构建对乙酰氨基酚(APAP)诱导的DILI模型。采用小鼠原代肝细胞和小鼠肝细胞系 (AML-12)进行体外实验。通过Ago 2相关RNA免疫沉淀结合高通量测序来识别miR-31的特异性靶点。
RNA免疫沉淀结合高通量测序的生物信息学分析发现,JNK信号上游分子细胞分裂周期蛋白42 (Cdc42)是miR-31的特异性靶点,可形成miR-31/Cdc42/磷酸化混合谱系激酶3 (p-MLK3)负反馈环,限制JNK过度活化。
通过Ago2 RIP-seq获取miR-31的靶标mRNA。
(A)Ago2 RIP-seq流程图。(B)miR-31靶标筛选策略。(C)RIP-seq中Ago2结合Cdc42的peak峰图。(D)APAP诱导DILI模型小鼠中,miR-31敲除后促进Cdc42的表达。(E)APAP诱导DILI细胞模型中,miR-31敲除后促进Cdc42的表达。(F)APAP诱导DILI模型小鼠中,JNK上游信号通路中p-MLK3和p-MLK4表达上调。(G)APAP诱导DILI细胞模型中,转染miR-31的inhibitor后,p-JNK以及JNK上游信号通路中p-MLK3和p-MLK4表达上调。(H)双荧光素酶报告基因实验验证miR-31与Cdc42靶向关系。(I)APAP诱导DILI细胞模型中,敲低Cdc42后p-JNK表达下调。(J)将I中细胞进行流式检测细胞坏死情况。
miR-31通过下调Cdc42抑制APAP诱导DILI肝细胞ROS/JNK/线粒体的过度激活的分子调控机制模型图。