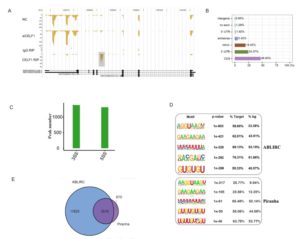
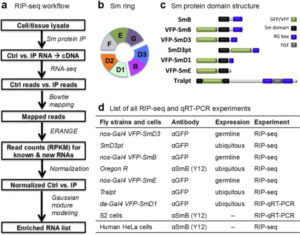
CELF1 preferentially binds to exon-intron boundary and regulates alternative splicing in HeLa cells
RNA结合蛋白(RBPs)参与到了转录后调控,包括RNA的可变剪接,RNA的空间折叠,从而导致RNA的表达量的变化。因此精确确定RBPs在RNA上的结合位置和结合特点是至关重要的。目前已经有成功解析RBP结合位点的方法,如CLIP-seq。但CLIP-seq也有很多的缺陷:如实验复杂,成功率低,文库的丰富度不够。和CLIP-seq比较类似的RIP-seq,虽然少了一些步骤,但获得的结果和CLIP-seq比较类似。最近有一项研究报道,使用DO-RIP-seq技术,也可以成功地获得RBPs的结合位点。而且能够获得更好的丰富度,这为RIP-seq分析RBPs的结合位点提供了方案和依据。