1.文章简介
| 文章题目 | Transcriptome analysis reveals the complexity of alternative splicing regulation in the fungus Verticillium dahliae |
| 中文题目 | 转录组分析揭示了在黄萎病菌中选择性剪接调控的复杂性 |
| 期刊名 | BMC Genomics IF:3.79 |
| 发表时间 | 2017 |
| 单位 | 生命之美 & 湖北农科院 |
| 实验材料 | 黄萎病菌两种菌株 |
| 测序平台 | Illumina Genome Analyzer IIx |
| 相关产品 | 转录组测序 |
2.研究背景
- 可变剪接是产生转录组多样性和蛋白复杂性的一种重要的转录后调控机制。然而,真菌中的可变剪接研究还不多。高通量测序发现真菌的内含子比之前预料的多,而且和植物一样,内含子保留是真菌中的主要剪接模式。
- 真菌中已经有一些可变剪接调控其生物学功能的例子。例如,黄瓜霜霉病菌中,PscRXLR1通过可变剪接调控可以从编码一个无功能的蛋白变成编码一个有功能的蛋白。玉蜀黍黑粉菌中,UmRrm75在细胞生长,致病性等方面的功能也受到可变剪接的调控。也有报道称有致病性真菌的毒力相关基因也受到可变剪接的调控。
- 黄萎病菌是一种可导致多种经济作物枯萎的真菌,而且缺乏有效的农药。黄萎病菌基因组的发布为从分子层面研究其致病性提供了可能性。
3.研究方法和主要发现
运用RNA-seq 技术构建了两种黄萎病菌的转录组,使用自研的生物信息学方法对可变剪接进行鉴定,定量,差异分析,以及功能通路分析:
1. 对黄萎病菌样本进行RNA-seq,分析发现大约50%含有intron的基因都存在可变剪接 ;
2. 除了发现大量的内含子保留事件,发现也存在超过1000种其他类型可变剪接事件;
3. 可变剪接基因富集在菌丝发育,孢子产生,信号反应等通路,暗示病菌可能通过可变剪接增加其编码生物学功能相关蛋白的可能性,也可能与其毒性相关。
4.研究结果
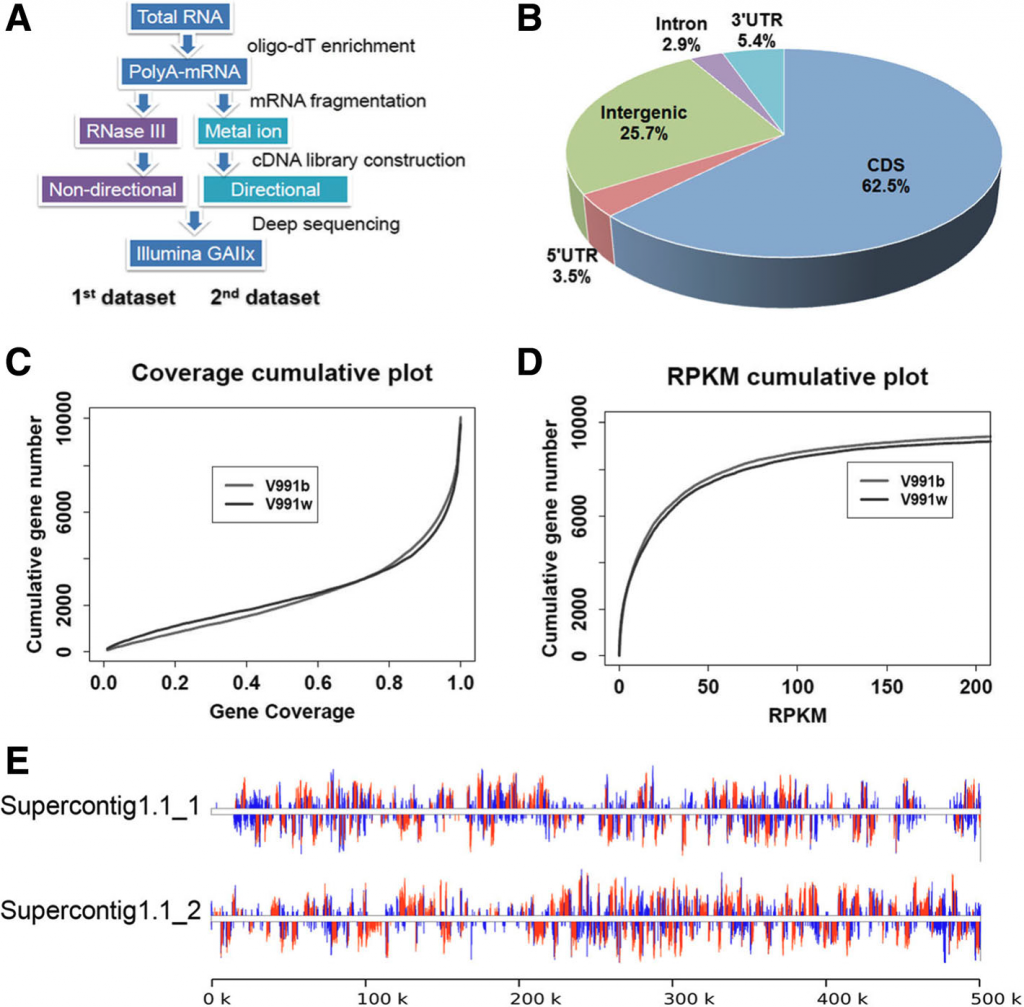
分析1:转录组质量控制和基础分析。首先对两个黄萎病菌(V991w和V991b)分离株进行了下一代测序,并对测序数据进行质量控制和基础分析,包括测序量,基因组不同区域的分布比例,基因检出数,序列覆盖度,序列覆盖率,序列深度,基因表达量分布等。
主要发现:使用polyA-mRNA的RNase III片段获得的结果发现5’和3’UTR区域的序列深度显着高于CDS区域,此结果可能是由于非编码区中富集的局部RNA二级结构引起的,该区域更易被RNase III消化,并在cDNA文库中选择性富集。使用离子片段化方法从相同的菌株构建了另一组cDNA文库,显示了CDS区域中序列深度的急剧增加。(图1)
分析2: 使用自主开发的内含子保留事件分析算法iRAS来系统鉴定转录组数据中的内含子保留可变剪接事件(IR)。验证:RT-PCR。
主要发现:黄萎病菌中存在大量内含子保留事件。这一部分首先对黄萎病菌的基因组的外显子和内含子特征进行了描绘,79.3%的蛋白质编码基因(8359)包含至少一个内含子, 19150个内含子占基因组序列的5.7%,内含子的平均长度为100 bp,含内含子的基因平均包含2.3个内含子。预示了真菌中存在着普遍的AS调控。接下来,文章使用自己开发的内含子保留事件分析算法iRAS来识别转录组数据中的内含子保留可变剪接事件(IR)。共找到1558个高可性的IR事件。RT-PCR验证了十几个事件。内含子的保留可能是由于原始转录物的聚腺苷酸化而没有剪接现有的内含子或基因中某些内含子而不是其他内含子的特异性保留。内含子保留可以通过破坏编码序列来改变基因的蛋白质编码潜能,并且还可以通过引入过早终止密码子(PTC)来干扰mRNA的稳定性。统计分析表明,有474个非最后一个内含子IR事件可能会导致PTC。(图2)
图2.黄萎病菌中内含子保留事件(IR)对生成mRNA的普遍调节。黄萎病菌基因的结构特征。显示了所有内含子(左)和外显子(中)的长度分布的统计数据,以及每个基因的外显子组成。通过当前iRAS算法从两个数据集中检测到的IR事件的RT-PCR验证(b)。我们开发了一种算法来可视化每个基因中的可变剪接事件。对于每个基因,将绘制映射到独特基因组位置的cDNA读图并编号,并指出替代剪接的类型和检测到AS的数据集。一个完整的例子显示在(b,左)中,电泳凝胶上方详细介绍了用于RT-PCR的含IR区域。为了简化表示并强调AS区域中的阅读布局,我们在顶部图上显示了包含AS事件的基因结构,已知外显子用青色框起来,内含子用蓝线框出,UTR区域用紫色框框出。 AS在下面放大,PCR引物的位置用黑色箭头表示,组成剪接产物的核苷酸长度用数字表示,其他外显子用绿色框出。黑色标度(水平轴)和下面的数字指示基因组位置。标尺下方显示了跨越剪接点的序列读数。红线和蓝线分别表示从V991b和V991w读取的数据。对于结读,未匹配的内含子区域以较细的绿线显示;完全对齐的读数如下所示。在少数情况下,会显示两个数据集的读取布局,并将第一个数据集的结果加框(c)。显示了对应于每对引物的RT-PCR验证结果的代表性电泳凝胶。两批总RNA用于RT-PCR验证; V991b和V991w的加载顺序不同。
分析3: 盒式内含子和内含子区内含子的鉴定和分析。
主要发现:文章中鉴定了几百个新的可变剪接模式:盒式内含子和内含子区内含子。盒式内含子的剪接导致mRNA编码的蛋白比未剪接的蛋白短,这些可变剪接可能会改变真菌基因产生不同的mRNA亚型。(图3)
图3. 涉及一对新的剪接位点的盒式内含子和内含子内含子。 a新鉴定的的外显子内含子。 其长度分布的统计信息(左); 这些内含子之一的RT-PCR验证(右)。 b涉及扩展UTR的外显子内含子。 c内含子内含子。 标签与图2中的相似。
分析4: 5’UTR和3’UTR注释。
主要发现:使用转录组数据对黄萎病菌基因组的5’UTR和3’UTR进行了进一步注释,注释了1591个新的5’UTR和2430个新的3’UTR,同时扩展了已有的146个5’UTR和78个3’UTR。
分析5: 使用aJAS算法鉴定非IR事件。验证:RT-PCR。
主要发现:文章开发了一种算法aJAS,对非内含子保留的其他7种剪接事件进行了鉴定和分析,包括可变5’剪接位点(A5SS),可变3’剪接位点(A3SS),盒式外显子(CE),跳跃外显子(ES),互斥外显子(MXE),互斥5’UTR(5pMXE)和互斥的3’UTR(3pMXE);以及两种混合类型包括可变的3’剪接位点和外显子跳过位点的结合(A3SS&ES),以及可变的天然5’剪接位点和外显子跳过位点的结合(A5SS&ES)。检出503个高可信的剪接事件,主要是A5SS(50%)和A3SS(35%)。使用RT-PCR对部分事件进行了验证。另外,在黄萎病转录组中也发现了不同剪接模式组合的复杂剪接事件,这说明真菌中的可变剪接比我们想象的更复杂。(图4)
图4. 复杂的可变剪接模式。 对黄萎病菌的5’和3’UTR的重新注释。 b在两个数据集的黄萎病菌V991b和V991w的转录组中,新鉴定的SJ的交集分析。 c-e涉及一个可变剪接位点的典型可变剪接事件的RT-PCR验证结果:可变3’剪接位点(A3SS); 可变5’剪接位点(A5SS)和Cassette Exon。 f涉及A5SS和外显子跳跃的组合可变剪接。 标签与图2中的相似。
分析6: 差异表达基因和差异剪接的关联分析。
主要发现:发现可变剪接事件没有明显影响mRNA转录水平。(图5)
图5. 选择性剪接与差异基因表达之间的相关性。 差异表达基因与内含子保留事件之间的相关性(a); 涉及一个可变剪接位点的可变剪接(b)。 在两个数据集中的每一个中识别出的V991b和V991w之间的差异表达基因中,都包含了至少一个可变剪接事件之一。 Left-1st数据集,Right-2nd数据集。 在一种菌株中检测到的AS事件在另一种菌株中检测不到,或者在两种菌株中检测到的AS事件均用不同的符号表示。 横轴(logMeanRPKM)表示每个基因的表达水平; 纵轴(logFC)表示基因表达水平在V991b和V991w之间的倍数变化。 每个点代表一个差异表达的基因
分析7: 可变剪接的功能通路分析
主要发现:该研究一共鉴定了5634个AS事件,位于4000多个基因中。通过对这些基因进行GO功能通路分析,包括多种生物学过程,表明AS机制可能调节真菌中大多数的分子活性和生物学功能。和表达差异基因所在的通路相比,可变剪接调控的基因更富集在菌丝体发育,多细胞器官的发育和解剖结构的发育。(图6)
图6. 黄萎病菌受可变剪接调控的生物学过程。 a由植物病原体中的可变剪接控制的生物学功能代表。 选择节点得分大于30的所有生物学过程term进行展示,并绘制相应term的节点得分。 星号表示优先受转录调控或选择性剪接控制的term。 b以整个基因组为背景,对备选剪接基因进行GO分析。 为了适应大小,将图分为两部分(左和右)
总结:
这项研究表明了选择性剪接在塑造低等真核生物的转录组和蛋白质组复杂性中的普遍作用。结合数十种复杂的可变剪接模式的鉴定,可变剪接机制在黄萎病菌中具有重要的作用。黄萎病菌不同AS模式的频率和分布与通过EST分析或转录组测序检测到的拟南芥和水稻的AS频率和分布惊人地相似,按高低排序为IR,A3SS,A5SS和ES。在可变剪接调控的基因的功能聚类表明,在真菌发育和繁殖,信号转导,线粒体软骨功能以及许多其他生物学功能(包括转录和转录后过程)中可变剪接发挥着关键作用。
参考文献
Lirong Jin1†, Guanglin Li2,4†, Dazhao Yu1*, Wei Huang1, Chao Cheng2, Shengjie Liao2,3, Qijia Wu3,5 and Yi Zhang2 (2017) Transcriptome analysis reveals the complexity of alternative splicing regulation in the fungus Verticillium dahliae. BMC Genomics (2017) 18:130

