RXBio Translates Sequence to Science and Industry
Tel: 027-87050299Email: sales@rxbio.cc
RXBio Translates Sequence to Science and Industry
RIP-seq研究的是自然状态下最原始的RBP和RNA的互作状态,捕获到的RNA未经酶切片段化,获得的RNA片段较长,也无需RBP切胶纯化步骤,操作流程简单,成功率高,但是不能得到RBP直接结合的RNA位点。
iRIP-seq(lmproved RlP-seq)是我司自主开发用于研究RBPs的新技术,将CLIP-seq技术中的紫外交联(UV-crosslinking)和MNase酶切实验加入到传统RIP技术中,同时用CLIP-seq数据分析流程获得RBP结合的peaks和Motif信息。该技术既准确识别了RNA结合位点,又保留了RIP-seq简单性。
iRIP-seq技术的优势在于其高效性和特异性,使其能够对研究目标生物系统中的RBP进行精准分析。此技术不仅可以成功检测到已知的RNA与蛋白间的相互作用,同时也能发现新的RNA结合蛋白以及它们参与的相互作用网络。此外,该技术还有助于我们揭示RNA修饰在转录和转录后调控过程中的重要性,深化我们对RNA和蛋白质相互作用的认知。总体来说,iRIP-seq为研究RNA和蛋白质之间的相互作用提供了强有力的支持,使我们能够更加深入地了解生物系统中RNA和蛋白质之间的复杂相互作用网络。这一理解不仅有助于揭示基因表达调控的奥秘,也为疾病机制的解析和药物的开发提供了新的视角和方向
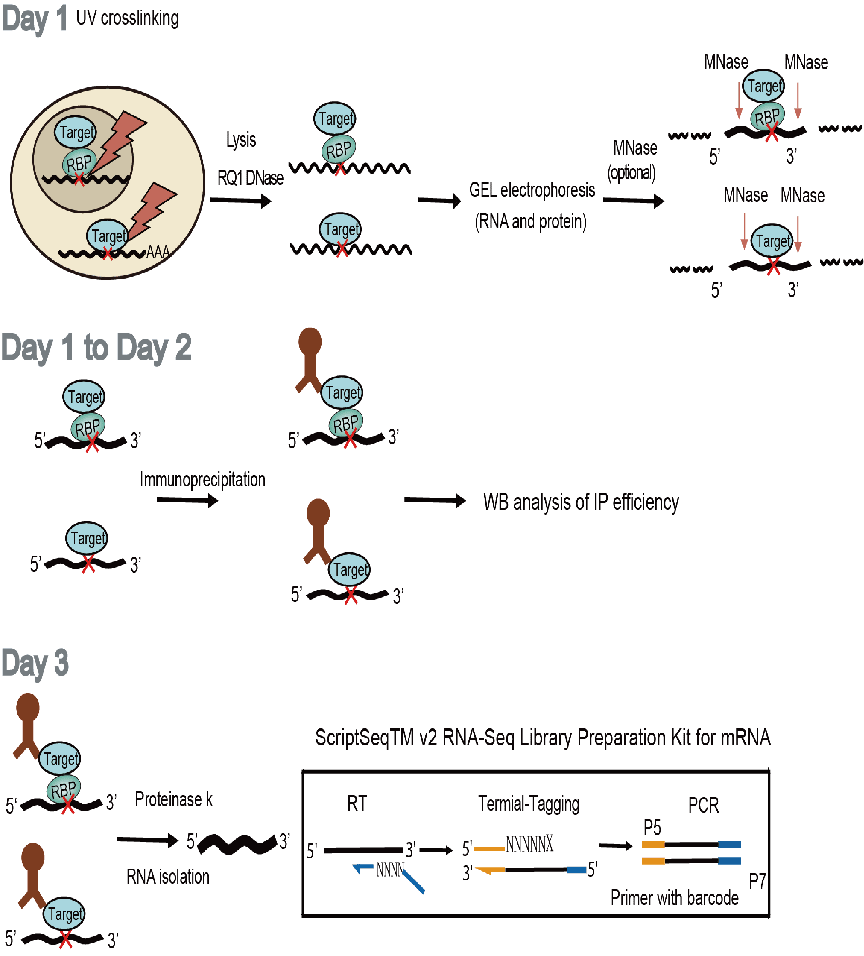
该技术的核心步骤包括增加的UV交联步骤,这一步骤通过产生共价键固定RBP-RNA互作位点,使得互作位点得到更稳定、更精准的识别。在进行酶切片段RNA的过程中,采用了MNase酶,这种酶具有钙离子依赖的特性,可以被EDTA等螯合剂终止酶反应。此外,更严格的离子去污剂和高盐漂洗条件使得实验过程中产生的噪音更小,提高了实验结果的信噪比。最后,改进的文库构建方法也是该技术的亮点之一,通过成功构建UV交联位点中断的RT产物文库,使得RBP-RNA互作位点的检测更为精准和高效
▶ FOXN3-NEAT1-SIN3A抑制复合物促进激素反应性乳腺癌的进展

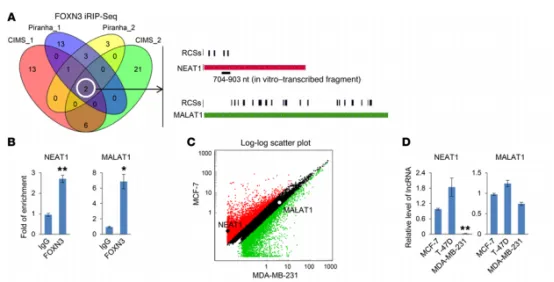
尚永丰院士团队发表于JCI(IF 15.9)上题目为“The FOXN3-NEAT1-SIN3A repressor complex promotes progression of hormonally responsive breast cancer”报道了转录因子FOXN3在乳腺癌转移中的作用及其作用机制。在MCF-7中采用改良RNA免疫沉淀偶联高通量测序(iRIP-seq)的方法鉴定了能够与FOXN3结合的RNA,经验证确定FOXN3与SIN3A复合体的相互作用需要一个受雌激素诱导的长链非编码RNA(lncRNA)NEAT1的参与。染色质免疫沉淀偶联高通量测序(ChIP-seq)结果和基于RNA靶标的深度测序捕获杂交分析(CHART-seq)数据联合分析证明,FOXN3/NEAT1/SIN3A复合体转录抑制包括GATA3和TJP1在内的一系列参与乳腺癌转移的重要基因。
▶ MEX3B通过下调上皮细胞TGFBR3 mRNA的稳定性抑制嗜酸性鼻息肉中胶原蛋白的产生

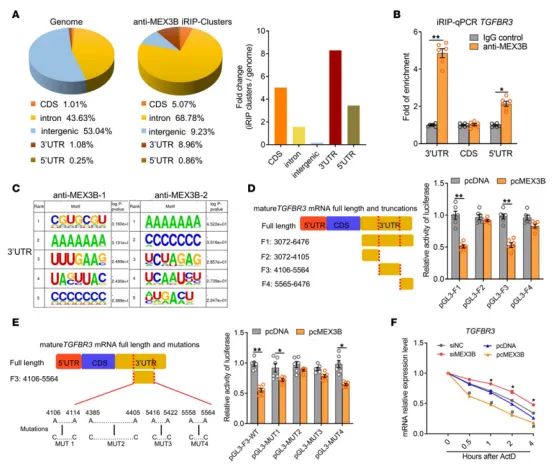
华中科技大学同济医学院附属同济医院副院长刘争教授团队在JCI insight(IF 8.0)上以期刊封面发表了题为“MEX3B inhibits collagen production in eosinophilic nasal polyps by downregulating epithelial cell TGFBR3 mRNA stability”的研究。为探讨MEX3B作为RBP在气道上皮细胞中的直接下游靶点,作者将 MEX3B 敲低和过表达后进行RNA-Seq和iRIP-Seq,共得到7个重叠基因,重叠的mRNA被认为是被MEX3B直接结合并调控的mRNA,其中TGFBR3尤为引人关注。TGF-βR3可以高亲和力结合并增强 TGF-β2 通路的功能, 在非嗜酸粒细胞性鼻息肉中 TGF-βR3 表达降低,引起 TGF-β2通路功能减弱,胶原家族相关基因的表达减少,促进组织水肿的形成。
▶ IGF2BP2通过稳定TSC1和PPARγ调节巨噬细胞表型激活和炎症疾病

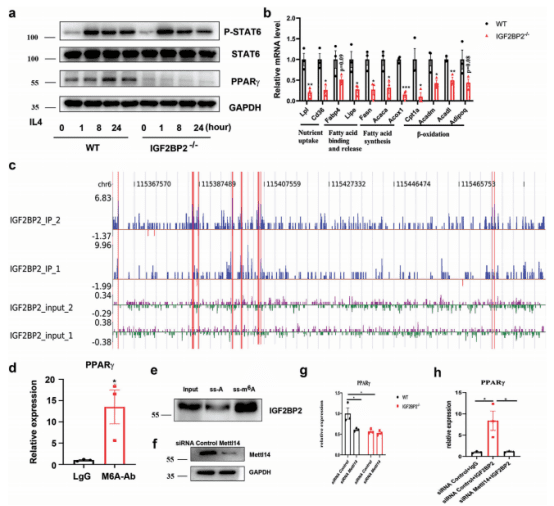
山东大学基础医学院李景新教授课题组在Advanced Science(IF 15.1)发表题为“The m6A Reader IGF2BP2 Regulates Macrophage Phenotypic Activation and Inflammatory Diseases by Stabilizing TSC1 and PPARγ”的研究论文。利用iRIP-seq技术发现巨噬细胞中IGF2BP2可结合Kcnn4、Arg1、Hist1h1b、Hist1h4c、Cd74、Ak2、H2-Eb1、Csf1r、PPARγ等多个在巨噬细胞激活及极化过程中发挥重要功能基因的mRNA,参与m6A修饰调控TSC1和PPARγ的表达。IGF2BP2通过TSC1- mTORC1通路和PPARγ介导的脂肪酸代谢使巨噬细胞从促炎表型向消炎表型倾斜。
▶ iRIP-seq技术探索了IGF2BP2在Jurkat细胞中的转录组范围的结合谱

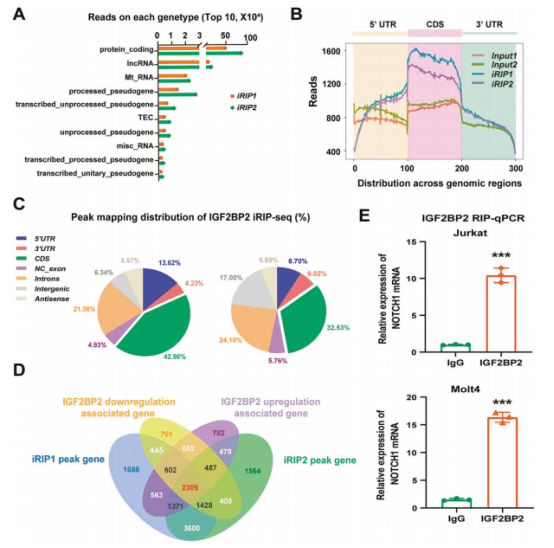
山东大学基础医学院李景新教授课题组又在Leukemia(IF 11.4)期刊发表一篇名为“Inhibition of the m6A reader IGF2BP2 as a strategy against T-cell acute lymphoblastic leukemia”的文章,利用iRIP-seq技术探索了IGF2BP2在Jurkat细胞中的转录组范围的结合谱,大多数IGF2BP2结合的reads位于mRNA中,并且在CDS中高度富集。然后对IGF2BP2上调或下调的Jurkat细胞进行RNA-seq测序,结合iRIP -seq数据鉴定出2309个调控基因,大量基因(NOTCH1、NKX2-1、BCL11B、RUNX1、FBXWT、STAT5B、DNM2、AKT1/2、MTOR、ABL1、DNMT3A和RPL11)与T-ALL发病相关。IGF2BP2作为m6A阅读器在甲基化过程中增加NOTCH1的稳定性,维持和增加转化T-ALL细胞的生长,IGF2BP2活性抑制剂JX5,有效抑制NOTCH1的激活和T-ALL的生长。
▶ 细菌中的A-to-I RNA编辑增加了对氧化应激的致病性和耐受性

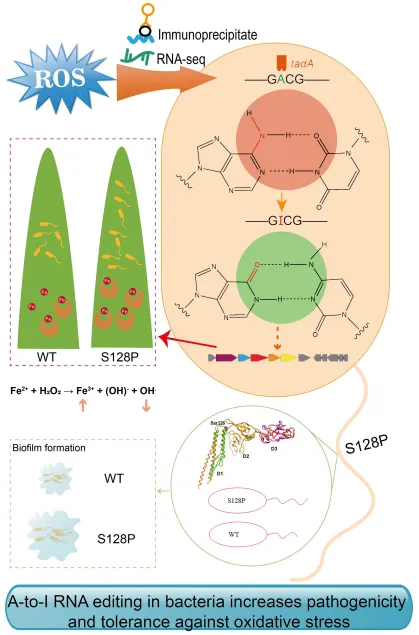
上海交通大学农业与生物学院朱勃/陈功友联合团队在PLoS Pathogens(IF 6.7)上发表研究成果“A-to-l RNA editing in bacteria increases pathogenicity and tolerance againstoxidative stress”,揭示了原核细菌通过A-to-1的 RNA编辑适应环境变化的新机制。研究团队通过iRIP-seq数据和自行开发的Python脚本对稻黄单胞菌稻生致病变种Xanthomonasoryzaepv.oryzicola(Xoc)在氧化应激下的A-to-1编辑事件进行识别和分析,发现A-to-|编辑在氧化应激胁迫下将鞭毛蛋白FliC第128位氨基酸由丝氨酸(S)变为脯氨酸(P),从而导致鞭毛丝结构的改变。这种转录后编辑可以通过增加生物膜的形成来增加氧化应激耐受性和和在水稻上的毒性。

电话:027-870502099
邮箱:sales@rxbio.cc
地址:武汉市东湖高新区高新二路388 号
光谷生物医药加速器 18 栋 1-2层
单细胞多组学 空间转录组
三代测序 功能基因组
表观遗传学 互作组学
单细胞大数据 数据深度挖掘

欢迎关注公众号「瑞兴生物」