RXBio Translates Sequence to Science and Industry
Tel: 027-87050299Email: sales@rxbio.cc
RXBio Translates Sequence to Science and Industry
DNA调控元件活性的一个重要特征在于其染色质可及性,也就是这些元件在染色质结构中的可接近性和可操作性。只有那些活跃的、具有活性的调节元件才有可能被细胞内部的机制所识别并进一步表达。因此,染色质的可及性,在转录调控的过程中,扮演着举足轻重的角色。它为研究转录因子如何通过调节染色质可及性来影响基因表达提供了关键线索。
2013年,美国斯坦福大学Howard Y. Chang研究团队开发了用于研究染色质可及性的方法ATAC-seq,其原理是通过转座酶Tn5容易结合在开放染色质的特性,然后对Tn5酶捕获到的DNA序列进行测序。ATAC-seq由于所需细胞量少,实验简单,可在全基因组范围内检测染色质可及性,已成为相关研究的首选技术方法。
✔ 起始量低:所需要细胞数10,000-,50,000个细胞,甚至可以做单细胞水平研究;
✔ 流程简单:步骤简单,成功率高;
✔ 特异性强:有效降低线粒体DNA数据污染;
✔ 重复性好:实验重复结果一致性好。
1.绘制开放染色质图谱,对于研究基因组结构和功能、疾病诊断和治疗等方面都具有重要的意义。
2.通过motif分析,识别参与基因调控的转录因子,对于理解基因表达的复杂机制以及与基因相关疾病的发生机制非常重要。
3.识别转录因子调控的靶基因和功能元件,研究转录因子在细胞生长、分化、凋亡等生命活动中的重要作用,也是理解基因表达复杂机制的重要途径。
4、结合核小体预测分析,精准地确定核小体的定位的规律和影响因素,为研究染色质结构和功能提供更全面的认识。
5.通过多组学联合分析获得基因表达与上游调控元件、转录因子的关联信息,解析转录因子靶向调控变化机制。
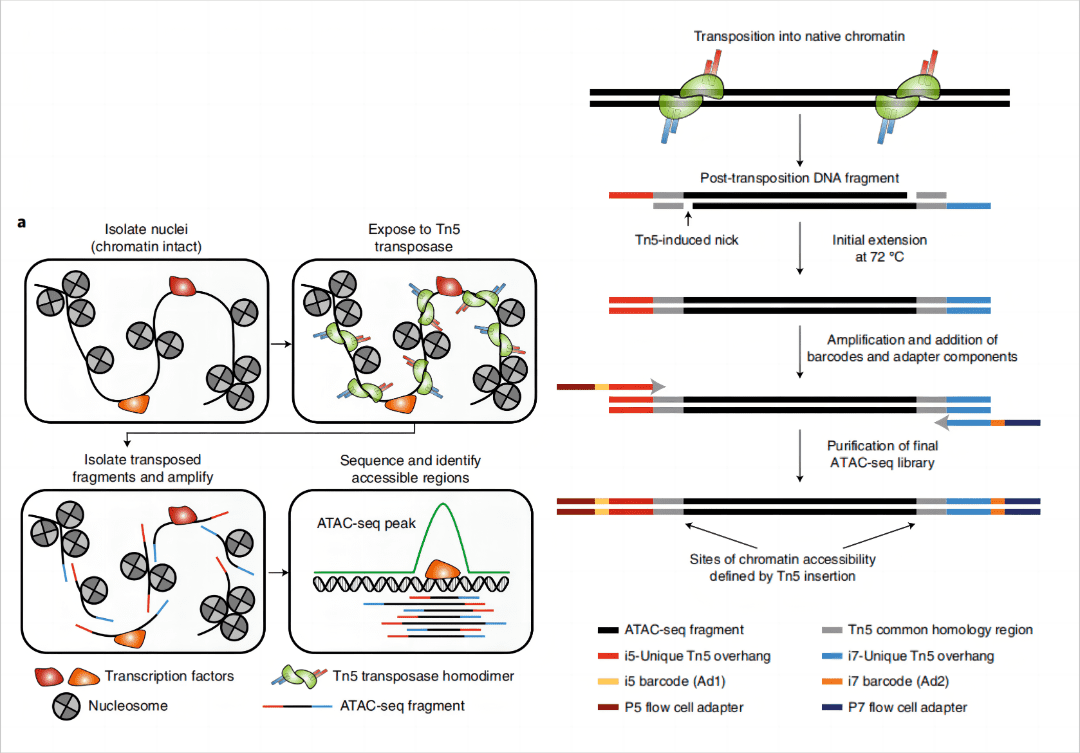
ATAC-seq的核心技术酶,Tn5转座酶可以将测序接头高效地插入到染色质可接近区域。然而,染色质的其它区域则与组蛋白紧密结合,以核小体的形式存在,形成一种紧密的结构,限制了转座酶的插入。Tn5转座酶能够精准地将测序接头直接插入到这些开放区的双链DNA中,对DNA进行片段化。随后,这些DNA片段经过提取和PCR扩增等后续处理,最终制备出适合二代测序的文库。通过测序和数据分析,我们可以进一步获得有关开放染色质的重要信息,这包括了转录因子(TF)的结合位点、组蛋白修饰位点等关键生物学信息。
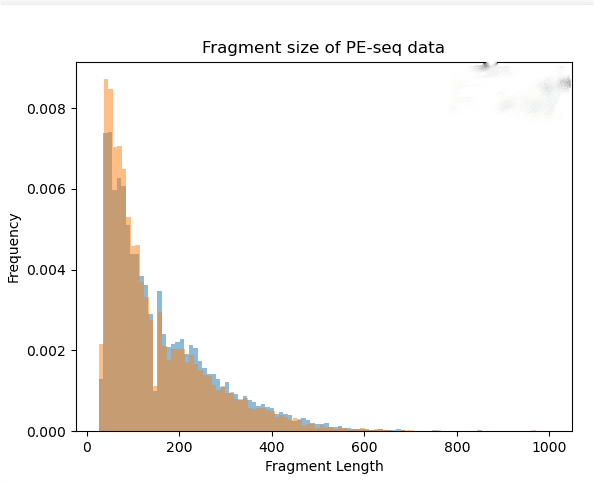
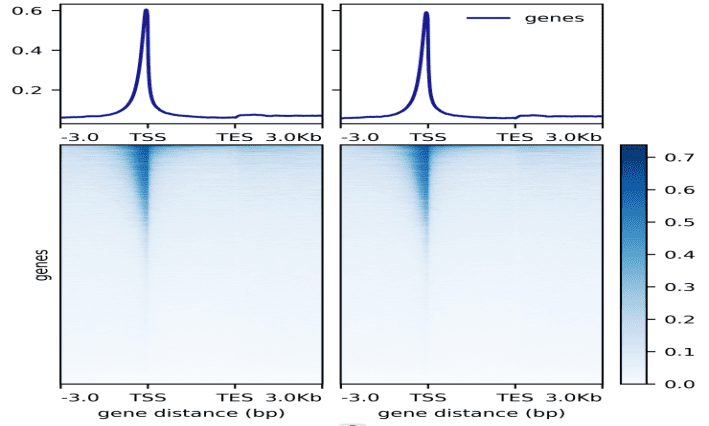
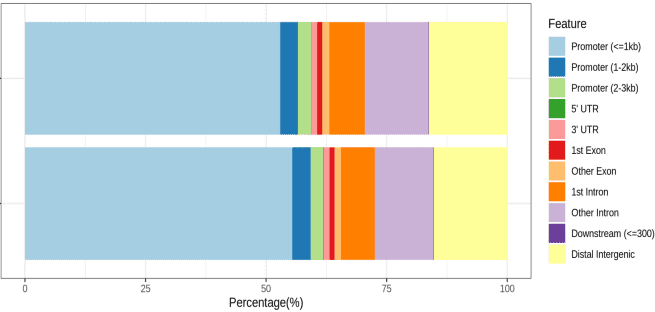
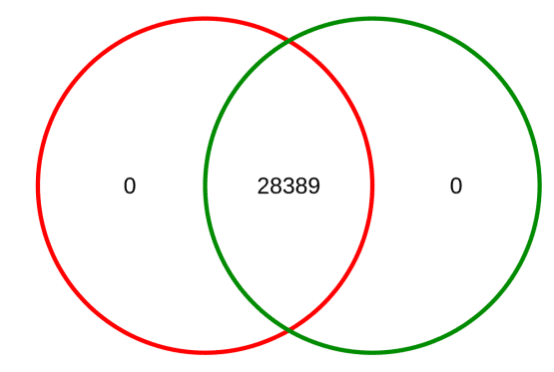
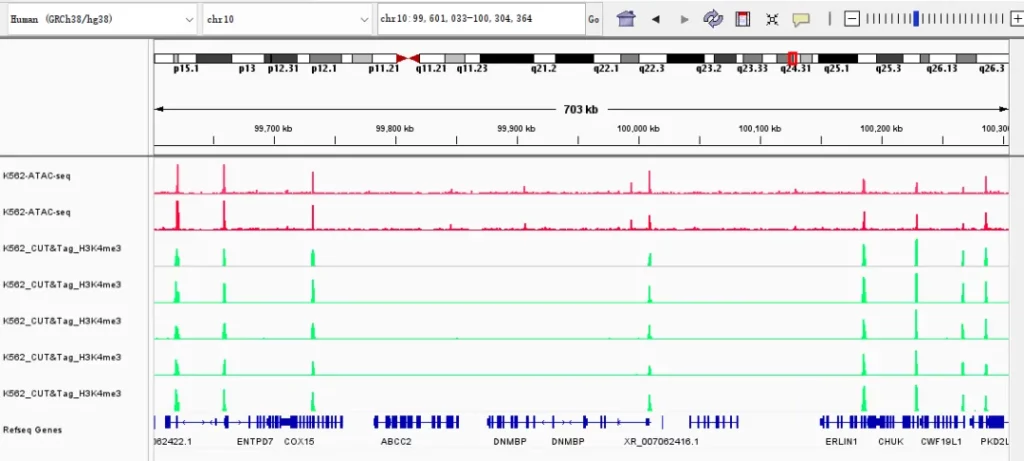
2018年10月26日,Howard Y. Chang教授团队在国际顶级学术期刊《Science》发表了染色质可及性研究的最新成果。利用ATAC-seq方法,结合癌症基因组图谱研究的现有数据,研究人员在410例囊括23种癌症类型的冷冻样本中,定位了562,709个可重复、转座酶可接近的染色质可及性位点。泛癌分析发现的染色质可及性峰值中,约有三分之二与过去发现的调控元件信息一致,表明ATAC-seq技术能够很好的重现过去的研究发现,同时还能发现大量新的染色质可及性敏感位点。
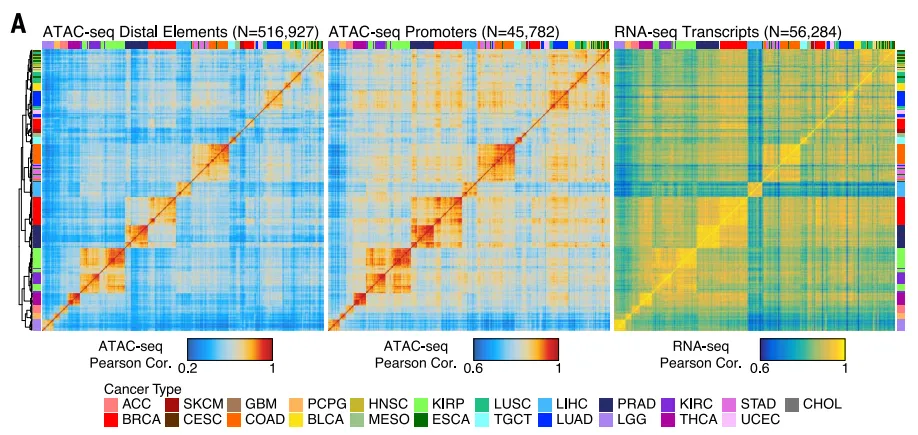
利用Distal elements和Promoters数据分型和RNA-seq数据分型的比较说明ATAC-seq数据可以用于区分不同肿瘤类型。
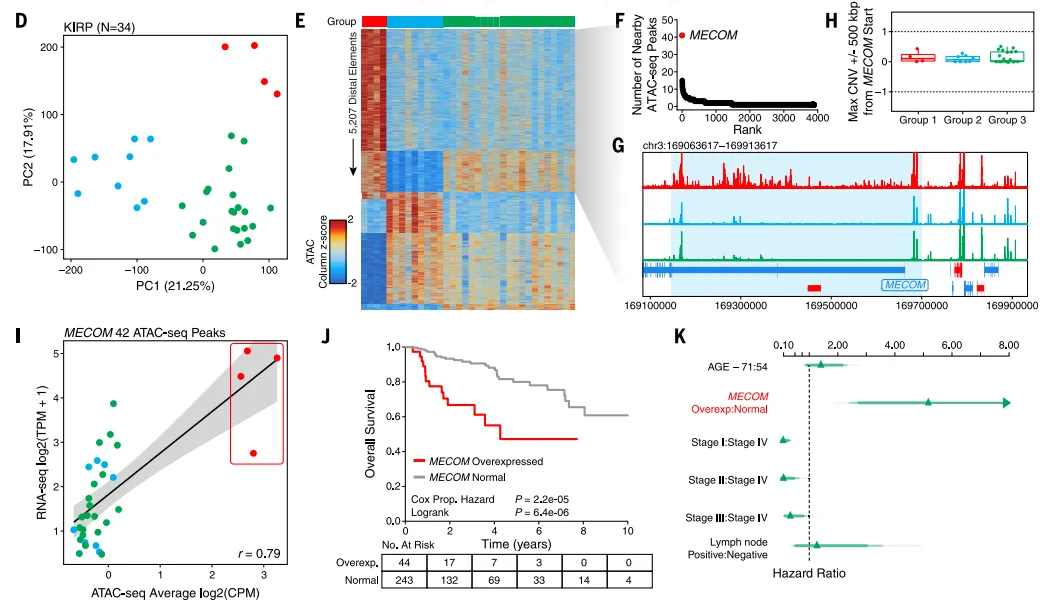
ATAC-seq数据对KIRP(一种肾癌类型)的分子分型研究发现图中红色代表了一种新的亚型,该亚型MECOM基因的染色质结合活性异常升高,这使得MECOM基因高表达。MECOM的过表达与较差的总生存率显著相关。

ATAC-seq数据对KIRP(一种肾癌类型)的分子分型研究发现图中红色代表了一种新的亚型,该亚型MECOM基因的染色质结合活性异常升高,这使得MECOM基因高表达。MECOM的过表达与较差的总生存率显著相关。
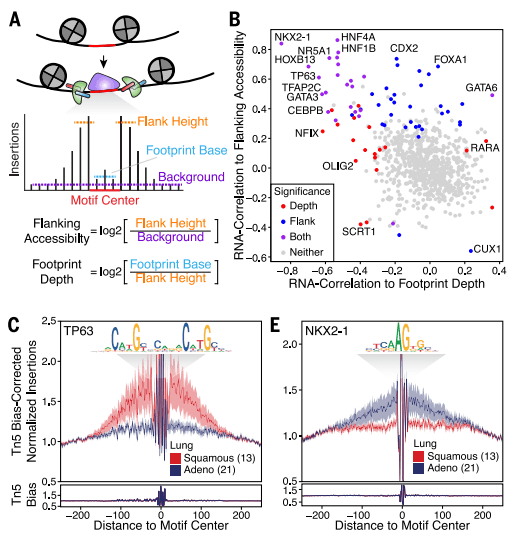
通过不同分型中特异性peak的寻找,发现这些特异性peak是以转录因子结合位点为主,并和甲基化密切相关,并发现了不同肿瘤类型特异的转录因子。
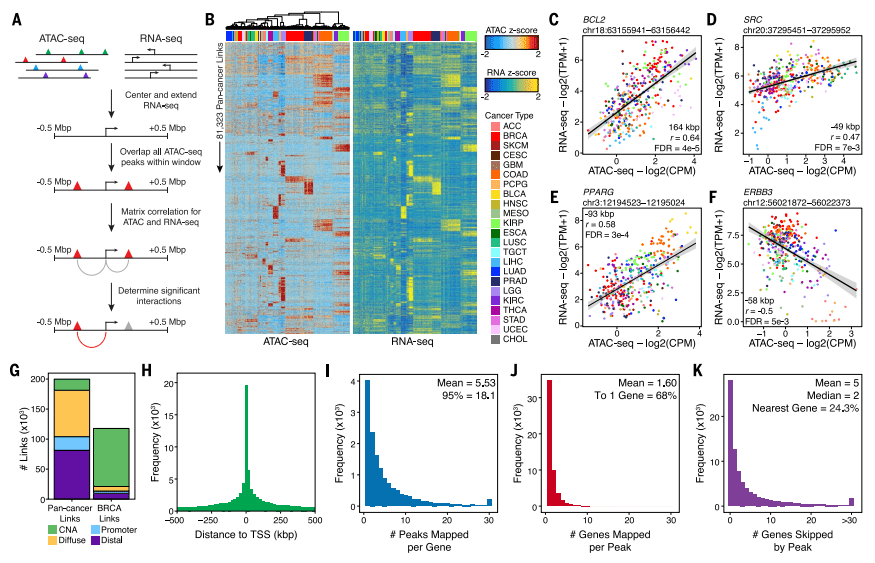
利用ATAC-seq数据和RNA-seq数据计算染色质可访问性与基因表达的相关性,一共预测了8552个蛋白编码基因表达调控中对应的peak。不同肿瘤类型中特异的peak不同。
参考文献
[1] Grandi F C, Modi H, Kampman L, et al. Chromatin accessibility profiling by ATAC-seq[J]. Nature protocols, 2022, 17(6): 1518-1552.
[2] M. Ryan Corces et al.,The chromatin accessibility landscape of primary human cancers.Science362,eaav1898(2018).DOI:10.1126/science.aav1898.

电话:027-870502099
邮箱:sales@rxbio.cc
地址:武汉市东湖高新区高新二路388 号
光谷生物医药加速器 18 栋 1-2层
单细胞多组学 空间转录组
三代测序 功能基因组
表观遗传学 互作组学
单细胞大数据 数据深度挖掘

欢迎关注公众号「瑞兴生物」