RXBio Translates Sequence to Science and Industry
Tel: 027-87050299Email: sales@rxbio.cc
RXBio Translates Sequence to Science and Industry
DNA甲基化是一种表观遗传修饰,由DNA甲基转移酶(DNMT)添加甲基团至胞嘧啶核苷酸,形成5-甲基胞嘧啶(5-mC),通常抑制基因转录。CpG岛是基因启动子区常见的区域,甲基化影响蛋白质结合和基因表达。研究者使用亚硫酸氢盐测序技术(BS-seq)和简化全基因组DNA甲基化测序(RRBS)技术来确定甲基化位点,BS-seq需要大量数据,而RRBS覆盖基因组约1-5%的CpG位点。
纳米孔测序技术能够直接检测胞嘧啶的甲基化状态(例如,在CpG位点),无需经过亚硫酸氢盐转化。CpG位点通常密集分布在被称为CpG岛(CGI)的区域,而大多数脊椎动物基因的启动子都位于这些CGI中。启动子内的甲基化模式变化与基因表达以及癌症等疾病状态密切相关:研究肿瘤样本与正常样本之间的甲基化差异有助于揭示肿瘤形成和发展相关的机制。自适应采样(AS)提供了一种快速、灵活且精确的方法,通过在测序过程中优先消耗非目标区域,从而增强对感兴趣区域(如cgi)的覆盖。RRMS协议使用户能够针对310 Mb的人类基因组进行测序,这些基因组富含CpG,包括所有注释的CpG岛、shores、shelves以及超过90%的启动子区域(100%的启动子包含超过4个CpG)。此外,还包括基因组中其他丰富的CpG区域。在.bed文件中记录的CpG位点总数达到718万个。
✔ 简单方便:不需要化学试剂转化,无需限制性内切酶处理。
✔ 覆盖度高:人靶向测序区域310 M,小鼠靶向测序区域308 M。
✔ 多种甲基化修饰:5mC、5hmC、6mA、4mC等。
变异分析:可以分析CNV、SV和SNP等相关信息。
✔ 单碱基分辨率:可以在单碱基水平检测。
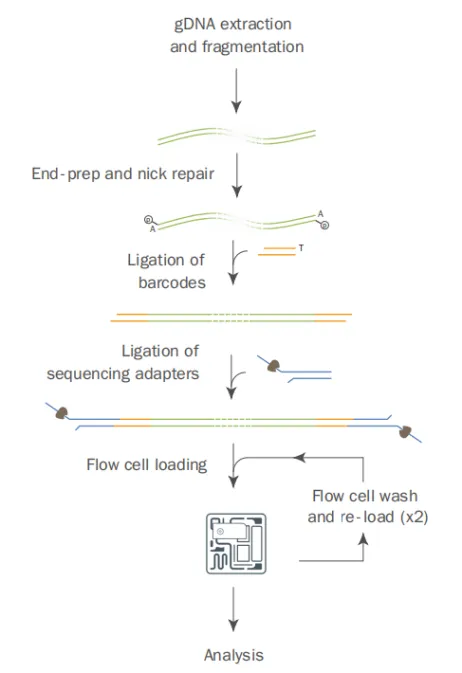
DNA-reduced representation methylation multiplex sequencing(RRMS)技术首先提取基因组DNA样本,对DNA进行片段化,打断后DNA平均长度在6kb。打断后的DNA末端进行修复,连接测序接头。在ONT测序仪器上面选择RRMS自适应采样测序程序。

详细信息请参考推文:送样规范 | 武汉瑞兴生物三代建库测序类相关产品送样取样建议
来自美国宾夕法尼亚大学的研究团队开发了长读长基因组DNA甲基化自适应采样测序的分析流程。该研究结果发表在Nature Communications上,文章题为“A signal processing and deep learning framework for methylation detection using Oxford Nanopore sequencing”。

牛津纳米孔测序能检测DNA甲基化,具有独特优势。自适应采样减少代表性甲基化测序,适用于CpG岛或印记区域。DeepMod2是一个深度学习框架,利用纳米孔测序的离子电流信号检测DNA甲基化。DeepMod2在CPU上高效运行,可推断表观单倍型或甲基化调用。与闭源工具Guppy和Dorado相比,DeepMod2性能相当。总之,DeepMod2是快速、准确的开源DNA甲基化检测工具。
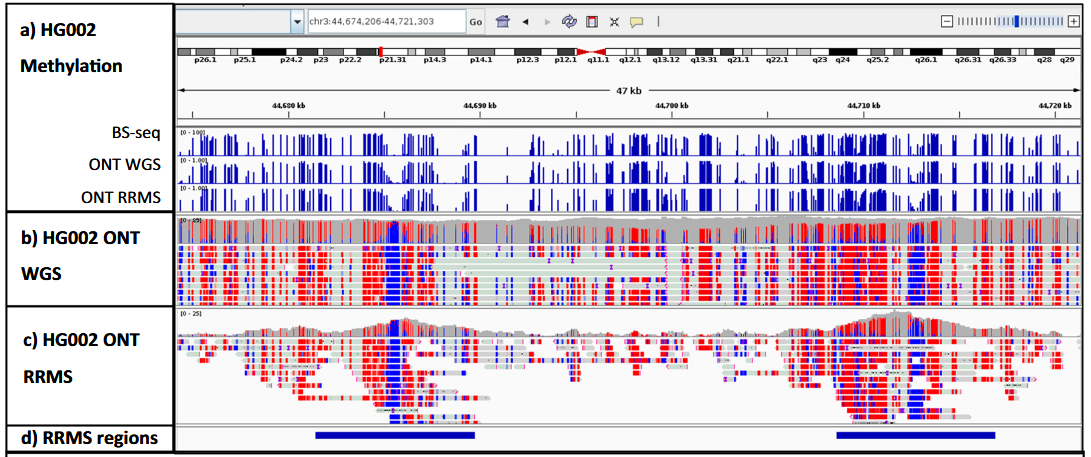
图展示了全基因组和RRMS纳米孔测序的reads和甲基化调用的IGV图,并比较了RRMS reads覆盖。使用DeepMod2,作者在HG002 RRMS中检测5mC,发现全基因组CpG基序内检测到39个胞嘧啶,靶区内检测到1399个。汇总后,全基因组范围内检测到248个CpG,靶区内检测到717个。RRMS区域检测到的聚合CpG数量多于搁浅CpG,后者数量不到前者的两倍。
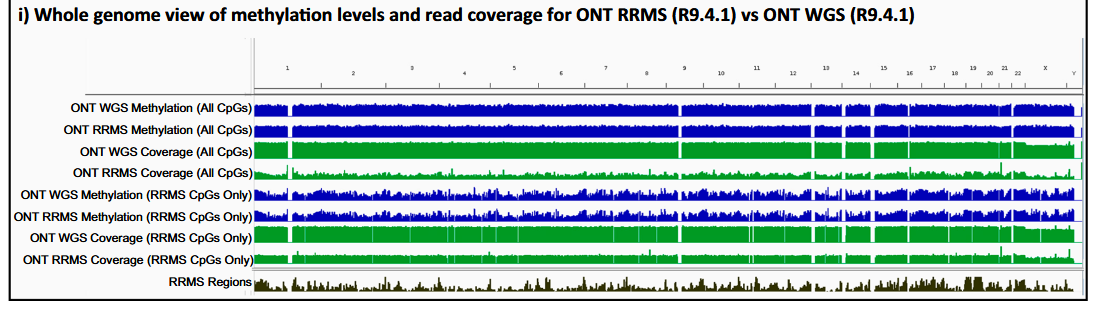
图展示了ONT RRMS和WGS数据集的覆盖范围和甲基化水平,以及RMMS CpGs与所有CpGs的轨迹和RRMS靶区轨迹。可观察到RRMS靶区相对于靶外区域的富集,如RRMS轨迹和覆盖峰的重合所示。
参考文献
【1】 Ahsan, M.U., Gouru, A., Chan, J. et al. A signal processing and deep learning framework for methylation detection using Oxford Nanopore sequencing. Nat Commun 15, 1448 (2024). https://doi.org/10.1038/s41467-024-45778-y

电话:027-870502099
邮箱:sales@rxbio.cc
地址:武汉市东湖高新区高新二路388 号
光谷生物医药加速器 18 栋 1-2层
单细胞多组学 空间转录组
三代测序 功能基因组
表观遗传学 互作组学
单细胞大数据 数据深度挖掘

欢迎关注公众号「瑞兴生物」