RXBio Translates Sequence to Science and Industry
Tel: 027-87050299Email: sales@rxbio.cc
RXBio Translates Sequence to Science and Industry
1958年,DNA双螺旋结构发现者弗朗西斯·克里克提出“中心法则”,阐述了DNA、RNA与蛋白质间的信息流动。基因转录产生前体RNA,经过可变剪接形成多种转录本,这些转录本翻译成不同的蛋白质异构体。选择性剪接使基因能产生多种蛋白质异构体,它们在结构和功能上可能有显著差异,并在特定疾病、组织或发育阶段中特异性表达。研究者们在《Nature Reviews Drug Discovery》上讨论了蛋白质异构体在药物发现中的作用,以及如何利用它们增强药物特异性和治疗效果【1】。因此,研究前体RNA的选择性剪接调节机制,以及这些转录本如何翻译成蛋白质并发挥功能,对于开发以蛋白质异构体为中心的治疗方法至关重要。
在人类基因组中,超过90%的基因会经历可变剪接,产生多种转录本。在短读长cDNA测序中,读段常比对到共有的外显子,难以确定来源。跨越外显子的接合点读段虽能提升分析精确度,但当转录异构体共享剪接位点时,效果受限。ONT-IrRNA-seq(Oxford Nanopore Technologies long-read RNA sequencing)能直接检测全长的isoform,捕捉独特转录本,消除或降低检测偏差,提高差异表达转录本分析的准确性。
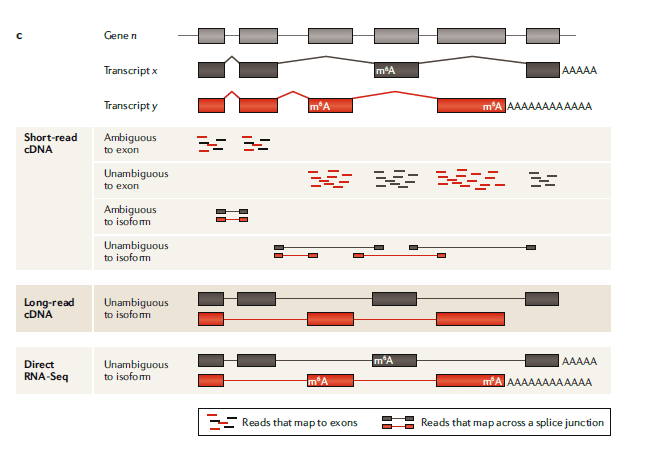
✔ 完善基因注释:发现新的转录本和基因,获得全面的转录组图谱。
✔ 差异分析:差异表达基因(DEG)、差异表达转录本(DET)、差异使用转录本(DTU)。
✔ 基因结构分析:准确分析可变剪接(AS)、多聚腺苷酸化位点(APA),3’UTR长度。
✔ 转录本分辩率:在转录本水平上解析基因表达的调控机制及其功能。
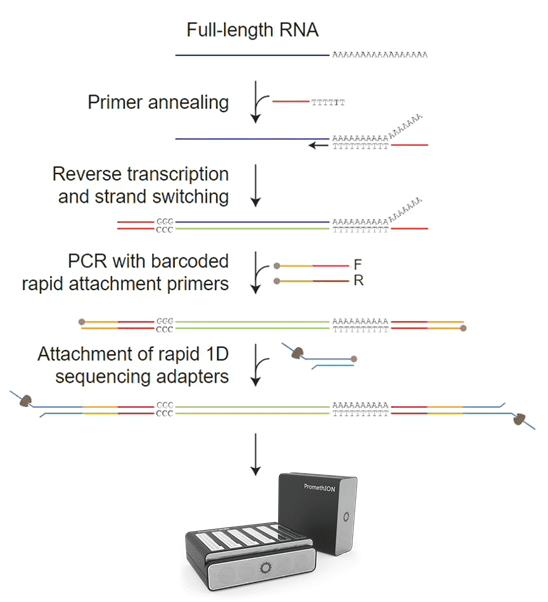
首先通过olig dT磁珠捕获获得含有polyA尾巴的RNA,接着使用olig dT+一段固定序列的引物逆转录合成cDNA第一链,然后把TSO序列通过链置换方式添加到cDNA 3’末端,使用引物PCR扩增形成双链cDNA,最后将双链cDNA末端进行修平和连接ONT测序接头形成测序文库。
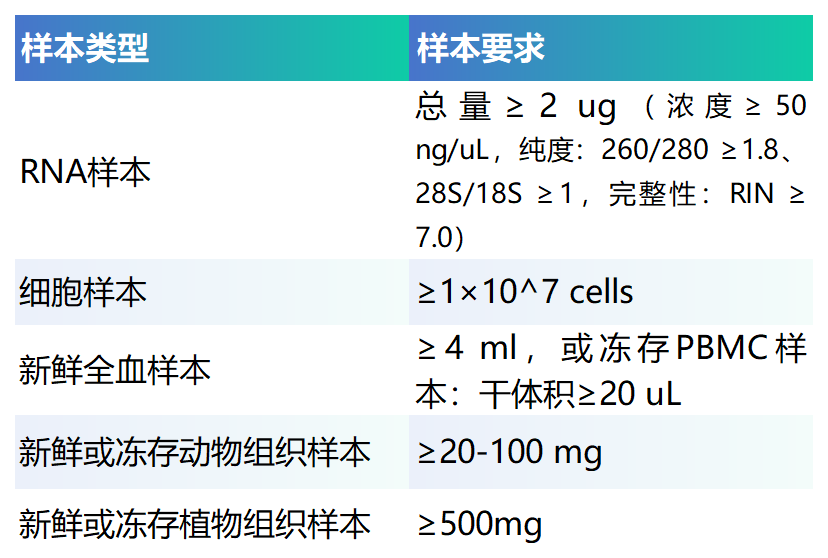
详细信息请参考推文:送样规范 | 武汉瑞兴生物三代建库测序类相关产品送样取样建议
Transcriptome Responses to Different Salinity Conditions in Litoditis marina, Revealed by Long-Read Sequencing
中文标题:长读长测序揭示 Litoditis marina 对不同盐度条件的转录组反应
期刊名:Genes
影响因子IF:2.8
样本:海洋线虫 Litoditis marina
实验分组:3‰、30‰和60‰盐度环境
技术方法:Oxford Nanopore Technologies (ONT) long-read sequencing
本研究利用ONT长读测序技术研究了L. marina在不同盐度下的转录组反应。在3‰、30‰和60‰盐度环境下得到了大量非冗余长读转录本,包括新转录本。研究发现,不同盐度条件下,氧化还原酶活性、离子转运等过程在ONT测序数据中均有所体现。随着盐度增加,特定基因表达量显著变化,GO富集分析显示细胞组分组装和辅酶生物合成过程在较高盐度下更为活跃。此外,研究还揭示了不同盐度下海洋线虫选择性聚腺苷酸化的模式,为理解其全盐性形成和演化机制提供了新视角,可能对研究气候变化引起的盐度变化有帮助。
Long-read RNA sequencing identifies regionand sex-specific C57BL/6J mouse brain mRNA isoform expression and usage
中文标题:长读长 RNA测序可识别区域和性别特异性 C57BL/6J 小鼠大脑 mRNA 亚型表达和用途
期刊名:Molecular Brain
影响因子IF:3.3
样本:C57BL/6J 小鼠
实验分组:小脑、皮质、海马体和纹状体组织
技术方法:Oxford Nanopore Technologies (ONT) long-read sequencing
选择性剪接(AS)是生物异质性的关键因素,与多种疾病相关。准确测量AS对于理解分子表型至关重要。长读长测序技术比短读长测序更精确,适合高通量实验。作者使用Oxford Nanopore Technologies (ONT)长读长RNA测序分析了小鼠大脑不同区域的AS性别差异。分析超过8500万个读数后,发现大脑各区域间及性别间的差异基因表达(DGE)、差异转录本表达(DTE)和差异转录本使用(DTU)均显著。特别是小脑与其他区域相比差异最大,皮层DTU的性别差异最明显。作者还观察到两种性别DTU模式,这可能解释神经和精神疾病性别差异的原因。最后,作者开发了一个Shiny Web应用程序供进一步研究。这项研究强调了AS在生物异质性中的作用和长读长测序在理解大脑AS中的应用价值。
Principal Component Analysis of Alternative Splicing Profiles Revealed by Long-Read ONT Sequencing in Human Liver Tissue and Hepatocyte-Derived HepG2 and Huh7 Cell Lines
中文标题:人肝组织和肝细胞来源的 HepG2 和 Huh7 细胞系中长读长 ONT 测序揭示的选择性剪接特征的主成分分析
期刊名:International Journal of Molecular Sciences
影响因子IF:4.9
样本:HepG2 和 Huh7 细胞
技术方法:Oxford Nanopore Technologies (ONT) long-read sequencing
ONT开发的长读长RNA测序能直接定量转录异构体,为替代剪接分析提供了一个合适的度量。利用主成分分析(PCA)作为工具,作者成功地将正常肝组织和肝癌细胞HepG2和Huh7的样本在二维空间中清晰分组。全转录组分析显示,无论是否包括所有基因,样本都能聚类。但在分析特定的药原基因时,仅包括表达基因的聚类效果变差。基于PCA,作者识别了对样本聚类贡献最大的基因子集,并通过基因本体分析确定了前20个与翻译和调控相关的主要生物过程。这个指标可作为现有指标的补充,特别是在长读测序的转录组研究中。
Full-Length Immune Repertoire Reconstruction and Profiling at the Transcriptome Level Using Long-Read Sequencing
中文标题:使用长读长测序在转录组水平上进行全长免疫库重建和分析
期刊名:Clinical Chemistry
影响因子IF:7.1
样本:8例急性淋巴细胞白血病患者、3例过敏性疾病患者、4例银屑病患者和5例前列腺癌患者的外周血
技术方法:Oxford Nanopore Technologies (ONT) long-read sequencing
免疫库多样性使得传统测序重建全长免疫库具挑战性。作者开发了基于线性滚环扩增和纳米孔测序的全长红外测序(FLIRseq)工作流程。FLIRseq优于BCR/TCR-seq,提供长CDR3、B细胞同种型和很少使用的V基因序列。FLIRseq观察到白血病缓解期患者克隆型多样性增加,异常BCR/TCR百分比下降。对于患有过敏性疾病或牛皮癣的患者,FLIRseq提供了有关V(D)J重组和特定免疫球蛋白类别的直接见解。FLIRseq能够对直接V(D)J重组和免疫球蛋白类别进行公正且全面的分析,有助于表征致病机制、监测微小残留病和定制过继细胞疗法。
Long-read sequencing reveals the landscape of aberrant alternative splicing and novel therapeutic target in colorectal cancer
中文标题:长读长测序揭示了结直肠癌中异常选择性剪接和新治疗靶点的概况
期刊名:Genome Medicine
影响因子IF:10.4
样本:76例结直肠癌组织和10例正常组织
技术方法:Oxford Nanopore Technologies (ONT) long-read sequencing
选择性剪接在致癌和癌症进展中关键。结合长读长测序与短读长RNA-seq研究CRC转录组复杂性,发现90,703个转录本,>62%为新颖。新转录本多具样本特异性,低表达,多外显子,癌基因特征明显。1472个差异表达的选择性剪接事件与CRC预后相关,新亚型或影响患者生存。TIMP1 Δ4-5在CRC中下调,抑制肿瘤生长和转移。SRSF1维持TIMP1外显子4-5包含。CRISPR/dCasRx策略可抑制CRC生长。新发现为CRC研究提供资源,TIMP1 Δ4-5或为潜在治疗靶点。
Transcriptome variation in human tissues revealed by long-read sequencing
中文标题:长读长测序揭示人体组织转录组变异
期刊名:Nature
影响因子IF:50.5
样本:88例 GTEx 组织和细胞系样本
技术方法:Oxford Nanopore Technologies (ONT) long-read sequencing
转录本结构调节产生多样性,对人类疾病有重要作用。作者使用 Oxford Nanopore Technologies 平台展示了 GTEx 组织和细胞系的 88 个样本的长读长 RNA-seq 数据集,补充了 GTEx 资源。共鉴定了 70,000 多个新转录本,并验证了 10% 的新转录本的蛋白质表达。开发了计算包 LORALS,分析罕见和常见变异对转录组的遗传影响,表征了等位基因特异性表达和转录本结构事件,为遗传变异引起的转录本改变提供新见解,并强调长读数据的分辨率。作者还发现 PTBP1 敲除后转录物结构扰乱,揭示了细胞环境改变的遗传调控效应。
参考文献
【1】 Kjer-Hansen, P., Phan, T.G. & Weatheritt, R.J. Protein isoform-centric therapeutics: expanding targets and increasing specificity. Nat Rev Drug Discov (2024). https://doi.org/10.1038/s41573-024-01025-z.
【2】Stark, R., Grzelak, M. & Hadfield, J. RNA sequencing: the teenage years. Nat Rev Genet 20, 631–656 (2019). https://doi.org/10.1038/s41576-019-0150-2.
【3】Glinos, D.A., Garborcauskas, G., Hoffman, P. et al. Transcriptome variation in human tissues revealed by long-read sequencing. Nature 608, 353–359 (2022). https://doi.org/10.1038/s41586-022-05035-y .
【4】Sun, Q., Han, Y., He, J. et al. Long-read sequencing reveals the landscape of aberrant alternative splicing and novel therapeutic target in colorectal cancer. Genome Med 15, 76 (2023). https://doi.org/10.1186/s13073-023-01226-y

电话:027-870502099
邮箱:sales@rxbio.cc
地址:武汉市东湖高新区高新二路388 号
光谷生物医药加速器 18 栋 1-2层
单细胞多组学 空间转录组
三代测序 功能基因组
表观遗传学 互作组学
单细胞大数据 数据深度挖掘

欢迎关注公众号「瑞兴生物」